Gelöste Aufgaben/COVI: Unterschied zwischen den Versionen
Keine Bearbeitungszusammenfassung |
Keine Bearbeitungszusammenfassung |
||
| (21 dazwischenliegende Versionen desselben Benutzers werden nicht angezeigt) | |||
| Zeile 1: | Zeile 1: | ||
[[Category:Gelöste_Aufgaben]] | |||
[[Category:Anfangswertproblem]] | [[Category:Anfangswertproblem]] | ||
[[Category:Matlab]] | [[Category:Matlab]] | ||
=Aufgabenstellung= | ==Aufgabenstellung== | ||
Als Ingenieure können wir die COVID-19-Epidemie genauso modellieren | Als Ingenieure können wir die COVID-19-Epidemie in Deutschland genauso modellieren wie technische Systeme. Wir brauchen dazu "Koordinaten" - also Messgrößen - die die quatitativen Größen des Krankheitsverlaufs erfassen. | ||
<onlyinclude> | |||
[[Datei:Flatten the Curve.png|200px|mini|Numerische Lösung der COVID-19 Pandemmie als Anfangswertproblem.|alternativtext=COVID19|links]] | |||
Gesucht ist hier ein phänomenologisches Modell für die Entwicklung der Anzahl der Individuen, die | |||
*ansteckbar "''a''", | *ansteckbar "''a''", | ||
| Zeile 11: | Zeile 14: | ||
*genesen "''r''" | *genesen "''r''" | ||
sind. Für jede dieser Koordinaten müssen wir | sind. | ||
</onlyinclude> | |||
Für jede dieser Koordinaten müssen wir "Bewegungsgleichungen" - also Differentialbeziehungen in der Zeit als [[Anfangswertprobleme|Anfangswertproblem]] - hinschreiben. | |||
Das nennen wir "Modellbildung". | Das nennen wir "Modellbildung". | ||
| Zeile 21: | Zeile 27: | ||
Das Modell ist nicht dafür gemacht, um daraus quantitative Schlussfolgerungen zu ziehen.}} | Das Modell ist nicht dafür gemacht, um daraus quantitative Schlussfolgerungen zu ziehen.}} | ||
Die algebraischen und Differentialgleichungen, die wir erhalten, sind dabei nicht das Ergebnis von Gleichgewichtsbeziehungen wie in der Technischen Mechanik. Wir begnügen uns statt dessen damit, Phänomene der Epidemie mit unserer Mathematik zu erfassen. | |||
Und so gehen wir vor: | Und so gehen wir vor: | ||
| Zeile 36: | Zeile 41: | ||
infiziert. | infiziert. | ||
== Lösung mit Matlab == | == Lösung mit Matlab<sup>®</sup> == | ||
{{MyCodeBlock|title=Header| | {{MyCodeBlock|title=Header| | ||
text=Wir lösen hier das Anfangswertproblem zu nichtlinearen Bewegungsgleichungen. | text=Wir lösen hier das Anfangswertproblem zu nichtlinearen Bewegungsgleichungen. | ||
| Zeile 56: | Zeile 61: | ||
{{MyCodeBlock|title=Declarations|text=Wir verwenden diese Parameter aus einer Tabelle | {{MyCodeBlock|title=Declarations|text=Wir verwenden diese Parameter aus einer Tabelle | ||
[[Datei:COVI-Excel-Eingabedatei.png|mini|EXCEL-Datei mit Parametern]] | [[Datei:COVI-Excel-Eingabedatei.png|left|mini|EXCEL-Datei mit Parametern]] | ||
und erklären unten, wie wir sie einsetzten.|code= | und erklären unten, wie wir sie einsetzten.|code= | ||
<syntaxhighlight lang="matlab" line start= | <syntaxhighlight lang="matlab" line start=11> | ||
cd 'C:\Users\abs384\OneDrive\Confluence Sources\COVI' | cd 'C:\Users\abs384\OneDrive\Confluence Sources\COVI' | ||
addpath('.\functions') | addpath('.\functions') | ||
| Zeile 72: | Zeile 77: | ||
{{MyCodeBlock|title=Equations of Motion| | {{MyCodeBlock|title=Equations of Motion| | ||
text=Die Bewegungsgleichungen schreiben wir für die Anzahl der Individuen an, | text=Die Bewegungsgleichungen schreiben wir für die Anzahl der Individuen an, | ||
* die infiziert sind (Koordinate "i") und | * die infiziert sind (Koordinate "''i''") und | ||
* die genesen sind (Koordinate "r"). | * die genesen sind (Koordinate "''r''"). | ||
Die Anzahl der Individuen, die sich noch anstecken können, ergibt sich dann aus | Die Anzahl der Individuen, die sich noch anstecken können, ergibt sich dann aus | ||
<math>a(t)= | :: <math>a(t)=n_0-i(t)-r(t)</math> | ||
Unsere "Minimal-Koordinaten" des Systems sind also i und r. | Unsere "Minimal-Koordinaten" des Systems sind also ''i'' und ''r''. | ||
Jetzt lassen wir unserer Fantasie freien Lauf:| | Jetzt lassen wir unserer Fantasie freien Lauf:| | ||
code= | code= | ||
<syntaxhighlight lang="matlab" line start=1 | <syntaxhighlight lang="matlab" line start=21> | ||
function dydt = covid19(t,y,dt,DS,n,a0,lambda) | |||
% COVID19 Evaluate the COVID19 ODEs | |||
% | |||
% last updated: 2020-03-24 | |||
% author: Andreas Baumgart | |||
% present state of pandemy | |||
q = y(end,:); | |||
% individuals left to be infected | |||
a = n - sum(q); | |||
% assign i=0 for t<0 | |||
if length(y)>DS | |||
i = y(end-DS,1); | |||
else | |||
i=0; | |||
end | |||
% recovery rate | |||
r = dt*lambda(2)*i; | |||
% infected % recovered | |||
dydt=[dt*lambda(1)*q(1)*(a-a0)-r, r ]; | |||
end | |||
</syntaxhighlight> | </syntaxhighlight> | ||
}} | }} | ||
| Zeile 88: | Zeile 116: | ||
{{MyCodeBlock|title=Solving| | {{MyCodeBlock|title=Solving| | ||
text= | text= | ||
1: Die Bewegungsgleichung für r | <h4>1: Die Bewegungsgleichung für ''r''</h3> | ||
Die Änderung der Anzahl der Genesenen ist proportional zu Anzahl der Infizierten. Man könnte man also | Die Änderung der Anzahl der Genesenen ist proportional zu Anzahl der Infizierten. Man könnte man also | ||
::<math>\displaystyle \frac{d}{dt} r(t) = \lambda_2 \cdot i(t)</math> | |||
anschreiben. Allerdings genesen zum Zeitpunkt t die Personen, die sich zum Zeitpunkt t-ΔT infiziert hatten. Dazwischen liegt gerade der gesamte Krankheitsverlauf mit der Dauer ΔT. Also schreiben wir | anschreiben. Allerdings genesen zum Zeitpunkt t die Personen, die sich zum Zeitpunkt t-ΔT infiziert hatten. Dazwischen liegt gerade der gesamte Krankheitsverlauf mit der Dauer ΔT. Also schreiben wir | ||
::<math>\displaystyle \frac{d}{dt} r(t) = \lambda_2 \cdot i(t-\Delta T)</math> | |||
und nennen ΔT die Genesungsdauer - oder in Begriffen der Schwingungslehre: Totzeit (engl.: "dead-time"). | und nennen ΔT die Genesungsdauer - oder in Begriffen der Schwingungslehre: Totzeit (engl.: "dead-time"). | ||
2: Die Bewegungsleichungen für i | <h4>2: Die Bewegungsleichungen für ''i''</h4> | ||
Die Änderung der Anzahl der Infizierten steigt mit der Anzahl der Infizierten i selbst und mit der Anzahl der ansteckbaren Individuen a. | Die Änderung der Anzahl der Infizierten steigt mit der Anzahl der Infizierten i selbst und mit der Anzahl der ansteckbaren Individuen a. | ||
| Zeile 107: | Zeile 134: | ||
Also wählen wir | Also wählen wir | ||
::<math>\displaystyle \frac{d}{dt} i(t) = \lambda_1 \;\; i(t) \cdot (a(t)-a_0) - \frac{d}{dt} r(t) \;\;\text{ mit } a_0 = \alpha\cdot n_0</math>. | |||
Beim Auftragen des Produkts i∙(a-a0) unten erkennt man gut, wie die Funktion für i=0 und (a-a0)=0 Null liefert - also die Zuwachsrate der Infizierten Null ist, wenn niemand infiziert ist oder die Anzahl der ansteckbaren Individuen Null ist. Dazwischen steigt die Funktion steil an: | Beim Auftragen des Produkts i∙(a-a0) unten erkennt man gut, wie die Funktion für i=0 und (a-a0)=0 Null liefert - also die Zuwachsrate der Infizierten Null ist, wenn niemand infiziert ist oder die Anzahl der ansteckbaren Individuen Null ist. Dazwischen steigt die Funktion steil an: | ||
[[Datei:COVI-Ansteckungsfunktion.png|left|mini|Die "Ansteckungsfunktion" des Modells]] | |||
Damit haben wir zwei Bewegungsdifferentialgleichungen, die wir in die Matlab-Funktion "covid19" schrieben. | Damit haben wir zwei Bewegungsdifferentialgleichungen, die wir in die Matlab-Funktion "covid19" schrieben. | ||
| Zeile 117: | Zeile 143: | ||
Die Bewegungsgleichungen machen wir dimensionslos mit | Die Bewegungsgleichungen machen wir dimensionslos mit | ||
::<math>t = \Delta T \cdot \tau</math> | |||
und erhalten als Bewegungsgleichungen | und erhalten als Bewegungsgleichungen | ||
::<math> | |||
\displaystyle \frac{d}{d\tau} \left(\begin{array}{c} | |||
i(\tau)\\r(\tau)\end{array}\right)=\left(\begin{array}{c} | |||
\Delta T\cdot\lambda_1\;\cdot\; i(\tau)\cdot (a(\tau)-a_0) - \displaystyle \frac{dr(\tau)}{d\tau}\\ | |||
\Delta T \cdot \lambda_2 \; \cdot \; i(\tau-1) | |||
\end{array}\right) | |||
</math>.| | |||
code= | code= | ||
<syntaxhighlight lang="matlab" line start= | <syntaxhighlight lang="matlab" line start=51> | ||
% limit of infection | |||
a0= n*alpha | |||
% | |||
% initial values in Mio. individuals | |||
y = [ 1E-3 0]; | |||
% | % time steps and "dates" | ||
dt=0.1; | |||
t=0:dt:50; | |||
% dead-time given in integration steps | |||
% | DS=int16(round(DT/dt)); | ||
% make lambda dim'less | |||
lambda = DT*lambda; | |||
% Eulers Method | |||
% | for j=2:length(t) | ||
y(end+1,:)=y(end,:) + covid19(t,y,dt,DS,n,a0,lambda); | |||
end | end | ||
</syntaxhighlight> | </syntaxhighlight> | ||
| Zeile 151: | Zeile 180: | ||
{{MyCodeBlock|title=Postprocessing| | {{MyCodeBlock|title=Postprocessing| | ||
text=.| | text= | ||
Die Ergebnisse plotten wir für verschiedene Werte von λ: | |||
[[Datei:COVI-result-01.png|left|mini|... für ''λ<sub>1</sub>'' = 0.016]] | |||
[[Datei:COVI-result-02.png|left|mini|... für ''λ<sub>1</sub>'' = 0.012]] | |||
Hier sieht man, wie unterschiedliche Ansteckungsraten zu unterschiedlich hohen Maximalwerten in den Infizierten ''i'' zu einem bestimmten Zeitpunkt führen. Und warum es sinnvoll ist, dieses ''λ'' niedrig zu halten.| | |||
code= | code= | ||
<syntaxhighlight lang="matlab" line start=1 | <syntaxhighlight lang="matlab" line start=71> | ||
% plot results | |||
hold on | |||
plot(t,y(:,1)) | |||
plot(t,y(:,2)) | |||
plot(t,n-y(:,1)-y(:,2)) | |||
hold off | |||
legend('i(t)','r(t)','a(t)') | |||
xlabel('t / tBez →') | |||
ylabel('Individuen / Mio →') | |||
</syntaxhighlight> | </syntaxhighlight> | ||
}} | }} | ||
[[Kategorie:Totzeit]] | |||
Aktuelle Version vom 7. November 2021, 16:45 Uhr
Aufgabenstellung
Als Ingenieure können wir die COVID-19-Epidemie in Deutschland genauso modellieren wie technische Systeme. Wir brauchen dazu "Koordinaten" - also Messgrößen - die die quatitativen Größen des Krankheitsverlaufs erfassen.

Gesucht ist hier ein phänomenologisches Modell für die Entwicklung der Anzahl der Individuen, die
- ansteckbar "a",
- infiziert "i" - mit und ohne Sympthome - und
- genesen "r"
sind.
Für jede dieser Koordinaten müssen wir "Bewegungsgleichungen" - also Differentialbeziehungen in der Zeit als Anfangswertproblem - hinschreiben.
Das nennen wir "Modellbildung".
| 🧨 Dies ist kein zuverlässiges Prognosewerkzweug: |
Hier geht es um die Modellierung
|
Die algebraischen und Differentialgleichungen, die wir erhalten, sind dabei nicht das Ergebnis von Gleichgewichtsbeziehungen wie in der Technischen Mechanik. Wir begnügen uns statt dessen damit, Phänomene der Epidemie mit unserer Mathematik zu erfassen.
Und so gehen wir vor:
Wir modellieren eine Grundgesamtheit von n0 = 80 Millionen Individuen.
Die Pandemie soll bei einer Durchseuchung von α=50% zum Stillstand kommen - die Individuen sind dann so weit voneinander entfernt, dass eine Ansteckung unwahrscheinlich ist.
Zum Zeitpunkt t0=0 seien von insgesamt i0 Individuen mit
infiziert.
Lösung mit Matlab®
Header
Wir lösen hier das Anfangswertproblem zu nichtlinearen Bewegungsgleichungen.
Diese Gleichungen haben wir nicht - wir entwickeln sie ad-hoc und achten nur darauf, zentrale Phänomene abzubilden. Eine Abbildung der realen Zahlen ist nicht beabsichtigt.
%*******************************************************
%* matlab script *
%* version: R2020a *
%* author: Andreas Baumgart *
%* last updated: 2020-03-25 *
%* ref: Technische Mechanik mit Computer *
%* description: COVID-19 Simulation *
%* no spatial resoultion *
%*******************************************************
Declarations
Wir verwenden diese Parameter aus einer Tabelle

und erklären unten, wie wir sie einsetzten.
cd 'C:\Users\abs384\OneDrive\Confluence Sources\COVI'
addpath('.\functions')
% load parameters
parameter = readtable('parameter.xlsx');
for row=1:length(parameter.name)
eval(strcat(char(parameter.name(row)),'=', num2str(parameter.zahl(row))))
end
Equations of Motion
Die Bewegungsgleichungen schreiben wir für die Anzahl der Individuen an,
- die infiziert sind (Koordinate "i") und
- die genesen sind (Koordinate "r").
Die Anzahl der Individuen, die sich noch anstecken können, ergibt sich dann aus
Unsere "Minimal-Koordinaten" des Systems sind also i und r.
Jetzt lassen wir unserer Fantasie freien Lauf:
function dydt = covid19(t,y,dt,DS,n,a0,lambda)
% COVID19 Evaluate the COVID19 ODEs
%
% last updated: 2020-03-24
% author: Andreas Baumgart
% present state of pandemy
q = y(end,:);
% individuals left to be infected
a = n - sum(q);
% assign i=0 for t<0
if length(y)>DS
i = y(end-DS,1);
else
i=0;
end
% recovery rate
r = dt*lambda(2)*i;
% infected % recovered
dydt=[dt*lambda(1)*q(1)*(a-a0)-r, r ];
end
Solving
1: Die Bewegungsgleichung für r
Die Änderung der Anzahl der Genesenen ist proportional zu Anzahl der Infizierten. Man könnte man also
anschreiben. Allerdings genesen zum Zeitpunkt t die Personen, die sich zum Zeitpunkt t-ΔT infiziert hatten. Dazwischen liegt gerade der gesamte Krankheitsverlauf mit der Dauer ΔT. Also schreiben wir
und nennen ΔT die Genesungsdauer - oder in Begriffen der Schwingungslehre: Totzeit (engl.: "dead-time").
2: Die Bewegungsleichungen für i
Die Änderung der Anzahl der Infizierten steigt mit der Anzahl der Infizierten i selbst und mit der Anzahl der ansteckbaren Individuen a. Sie reduziert sich um die Änderung der Anzahl der genesenen Individuen r. Allerdings weiß man aus Erfahrung, dass eine Epedemie endet, wenn ein bestimmter Durchseuchungsgrad α erreicht ist. Also wählen wir
- .
Beim Auftragen des Produkts i∙(a-a0) unten erkennt man gut, wie die Funktion für i=0 und (a-a0)=0 Null liefert - also die Zuwachsrate der Infizierten Null ist, wenn niemand infiziert ist oder die Anzahl der ansteckbaren Individuen Null ist. Dazwischen steigt die Funktion steil an:
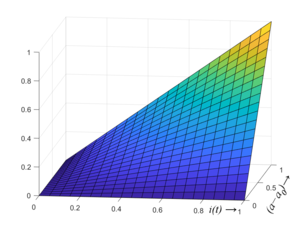
Damit haben wir zwei Bewegungsdifferentialgleichungen, die wir in die Matlab-Funktion "covid19" schrieben.
Die Bewegungsgleichungen machen wir dimensionslos mit
und erhalten als Bewegungsgleichungen
- .
% limit of infection
a0= n*alpha
% initial values in Mio. individuals
y = [ 1E-3 0];
% time steps and "dates"
dt=0.1;
t=0:dt:50;
% dead-time given in integration steps
DS=int16(round(DT/dt));
% make lambda dim'less
lambda = DT*lambda;
% Eulers Method
for j=2:length(t)
y(end+1,:)=y(end,:) + covid19(t,y,dt,DS,n,a0,lambda);
end
Postprocessing
Die Ergebnisse plotten wir für verschiedene Werte von λ:
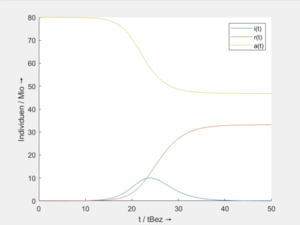
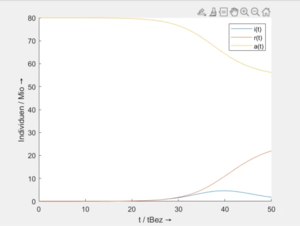
Hier sieht man, wie unterschiedliche Ansteckungsraten zu unterschiedlich hohen Maximalwerten in den Infizierten i zu einem bestimmten Zeitpunkt führen. Und warum es sinnvoll ist, dieses λ niedrig zu halten.
% plot results
hold on
plot(t,y(:,1))
plot(t,y(:,2))
plot(t,n-y(:,1)-y(:,2))
hold off
legend('i(t)','r(t)','a(t)')
xlabel('t / tBez →')
ylabel('Individuen / Mio →')